读Modern Statistics for Modern Biology随记
2019-09-15
Chapter 1 Introduction 引言
1.1 缘起
Modern Statistics for Modern Biology这本书,是斯坦福大学Susan P. Holmes 教授和 欧洲分子生物实验室的Wolfgang Huber 教授共同编写的。两位都是大牛,追他们的文章可以发现学科发展趋势的那种。
这本书2018年才出版,全文上网,还有配套的练习代码、数据,是很新鲜的学习材料。Holme教授在斯坦福大学暑期学校也教这门课,课程时长才十来天。我本想找找有没有视频跟着学学,不过一直没找到,只好跟着书学,并在这里写一些记录。
1.2 原书目标
The aim of this book is to enable scientists working in biological research to quickly learn many of the important ideas and methods that they need to make the best of their experiments and of other available data.
原书的目的是让生命口的工作者很快地学会能用于其实验和数据的重要理念和方法,这也是我想获取的技能。感觉强大的数据分析能力是一把手术刀,能突破学科壁垒,快速了解关键问题,并产出相对专业的成果;又如同包装设计师,一样的研究结果,用一些更加深入的统计工具来分析,或者以更加直观的统计图来呈现,会让人更有好感,更值得信赖、更容易接受。好像同一个小伙,了解女孩想事儿的套路了,则什么类型的姑娘都容易聊起来;再好好捯饬下自己,更容易获得她们的欣赏……大雾。
学习本书需要什么样的知识背景呢?我的感觉,基本生命专业的本科生都可以,有实际需求的最好。因为它并不涉及系统的统计背景知识,主要是对工具使用方法的学习,说白了就是培训“工匠技能”,有点数学基本知识、生物学背景再了解点R语言的皮毛,上就是了。不太熟的,上多了也就熟了。
所以,解决实际问题,也是本人的学习导向。首先要明确解决的问题是什么。有时候把问题定义清楚了,也就解决一半了。定义问题和解决问题的过程包括:
- 知道哪些前提数据
- 前提数据的表现形式、存储格式等
- 要分析的目标、方向
- 使用的工具(模型、包、函数)
- 结果的意义、表现形式(图、表、显著性等)
知道前提数据,并了解它们的表现形式,是很重要的环节。Susan在书中提出应对数据的差异性(The challenge: heterogeneity),也说明生命学科不同细分领域产生数据的多样性和差异性令人挠头。当然R语言作为一种语言,应对这种差异性再合适不过——语言嘛,见人说人话见鬼说鬼话就是了,前提是我们要大致了解这些神神鬼鬼,并学会一定的说话套路。
1.3 统计江湖的套路
首先作者放着这么一张图,统计学经典流程:做出原假设、实验检验、计算显著性(p值)、得出结论(原假设是否可靠)。并在图上有所创新,把原假设概念拓展到模型的宽度。

Figure 1.1: 这是Fisher提出的原假设检验过程。本书中可以把Hypothesis H0,想象成要拟合的模型,分析过程类似
然而这个图并不能涵盖本书内容,只是出来露个脸,可能作为一个统计的书,不提一下这个经典的理论有点缺乏正义性吧。
然后谈及对统计工作的划分角度,从大牛的视角开始(Tukey 1977),曾经分成这么两类:
- 探索分析 exploratory data analysis (EDA) 。
- 实证分析 confirmatory data analyses (CDA)。
原文中有对这两种方法的解释,但感觉说的不清不楚的,下面这个比较清晰 (Karageorgiou 2011)。
Exploratory Data Analysis (EDA) and Confirmatory Data Analysis (CDA) are two statistical methods widely used in scientific research. They are typically applied in sequence: first, EDA helps form a model or a hypothesis to be tested, and then CDA provides the tools to confirm if that model or hypothesis holds true.
基本上,EDA好像公安,遇见案子了要明确问题、搜集线索、整理证据、得出推论,而CDA就是检查院,要对公安得出的结论,以及整个证据链,进行检验,明确是否接受这个结论。接受了就送交法院,不接受呢,就得打回去,重新办理。
1.4 统计的常见概念和生命学科特殊性
原书中在EDA和CDA后,直接介绍生命学科搞统计遇到的的大 p 小 n 问题,介绍其如何不利于建模和得出推论。这里稍微扩展一点,明确一点概念和普通的建模,便于后面对照理解。
1.4.1 关于 p 和 n
先明确一下统计学常见的 p 和 n 是什么。 统计分析之前要有数据整理的过程,一般要把原始数据按 case/observation/个体/测量对象 和 variable/属性/变量 按照行和列进行整理。整理好的数据,有多少个case/个体(测量对象),n 就是多少;有多少variable/属性(变量),p 就是多少。收集数据的时候也要有这个意识,好的原始数据结构会减少后续整理的麻烦。
Figure 1.2: Excel中的示例,行为case,列为variable,这样放能直接应用数据透视表
1.4.2 一个普通的建模
从根上讲,所谓建模,其实就是找到要分析数据的,对我们有意义的某些方面的规律,并用公式表达出来;有时候需要把提取出来的规律应用到新的数据,得出预测的结果。Hadley把建模分为2类 (Grolemund and Wickham 2019) 才发现这本书Hadley居然不是第一作者:
| 模型类别 | 别称1 | 别称2 |
|---|---|---|
| * 预测模型 | “predictive” models | supervised models |
| * 数据分析模型 | “data discovery” models | unsupervised models |
预测模型好理解,我做个线性回归,这个回归模型肯定能在一定范围内预测趋势。可数据分析模型到底是啥玩意,咱也不知道,咱也搜不着,data discovery 从搜索结果看指向很宽泛的数据分析领域,unsupervised model呢,又和deep learning 经常出现在一块。所以我可以先把这个概念放一边——肯定不是啥严谨的东西,估计是主成分分析、聚类分析等不能外推规律的方法。 Hadley 书中也只介绍预测模型,下面摘一个简单的例子。
| x | y |
|---|---|
| 1 | 4.199913 |
| 1 | 7.510634 |
| 1 | 2.125473 |
| 2 | 8.988857 |
| 2 | 10.243105 |
| 2 | 11.296823 |
| 3 | 7.356365 |
| 3 | 10.505349 |
| 3 | 10.511601 |
| 4 | 12.434589 |
| 4 | 11.892601 |
| 4 | 14.257964 |
| 5 | 19.130050 |
| 5 | 11.738021 |
| 5 | 16.024854 |
| 6 | 13.273977 |
| 6 | 15.955975 |
| 6 | 16.894796 |
| 7 | 20.085993 |
| 7 | 17.171850 |
| 7 | 19.936309 |
| 8 | 21.725903 |
| 8 | 18.390913 |
| 8 | 22.475553 |
| 9 | 26.777010 |
| 9 | 22.805110 |
| 9 | 21.128305 |
| 10 | 24.968099 |
| 10 | 23.346422 |
| 10 | 21.975201 |
这组数据拿到手里,要建模,先做个图看看。
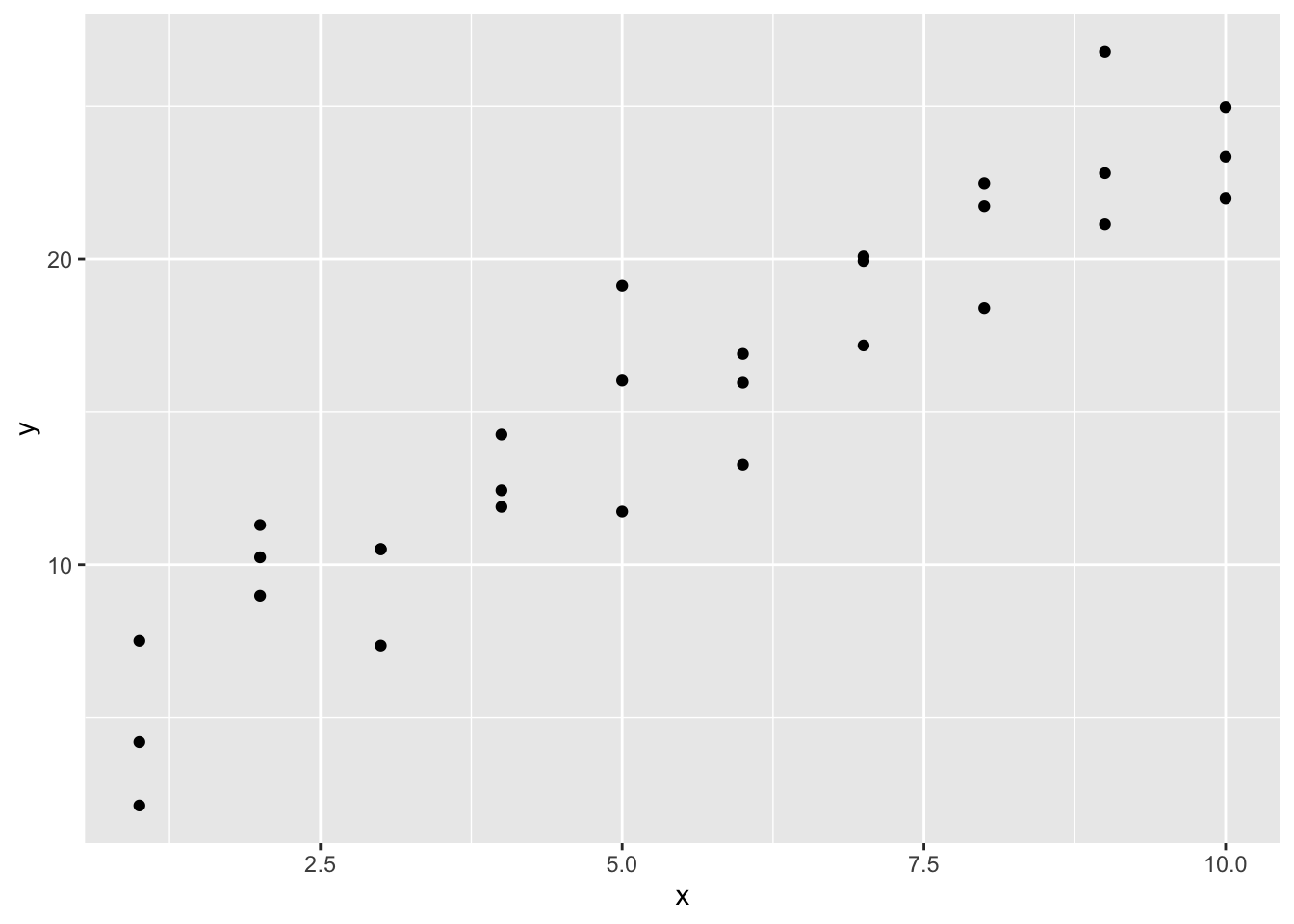
Figure 1.3: 拿眼一看就有明显的线性关系
然后要找到这个线性关系。高中数学告诉我们可以用 的形式表现这个线,然后就要找到 和 。实现方法很简单:
## (Intercept) x
## 4.220822 2.051533得到线性模型 。 如果用图形表现,可以这样实现。
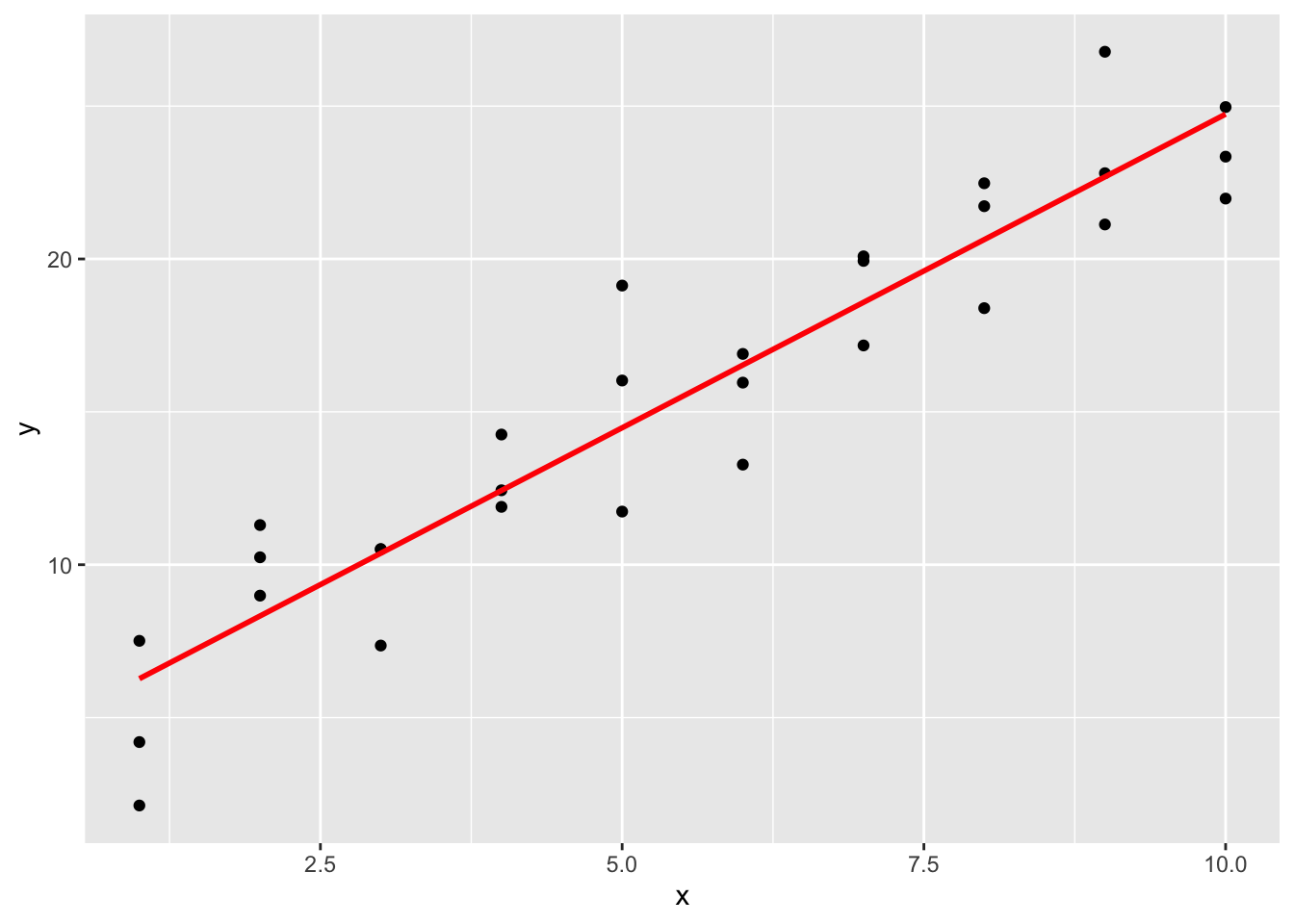
Figure 1.4: 线性模型的图形表现
这样一个简单模型就出来了,它充分归纳了已有数据的规律,也能推测数据之外的一定范围,比如可以推测11岁的时候大概有多重,这也是统计的目的所在——通过已知,预测未知。这里要注意,本数据中 p 是很少的(就俩, 和 ), n 是很多的,有30个,要建立模型,是需要确定 p 之间的系数的。
1.4.3 生命学科的大 p 小 n 问题
生命学科由于其特点,经常呈现出和刚才举例完全相反的数据。比如200个病人的基因样本,可能每个病人都有20000个基因。想想刚才的 ,如果想建立线性模型,理论上这个公式要扩充到20000个参数。
统计学家目前通过两种方向解决这个问题:
- 人工筛选变量(sparsity principle),把没用都的变量系数提前定义成0,或者接近0 (Hastie, Tibshirani, and Friedman 2016)。
- 贝叶斯经验方法(empirical Bayes),是sparsity principle的扩展(generalization),该方法认为“组间,或者性状之间,总有某些,甚至全部变量应该是相同或类似的”,然后发展出一系列方法,详细了解可参考 Speed (2011) 。
此外,原书中模拟(Simulations)的内容比较多。通过模拟一些数据练手,摆脱已有分析套路的局限,磨好屠龙刀,争取能更贴近实际的问题。
1.5 原书中主要内容
基于上述问题和发展出来的分析方法,原书中均有涉及,处理数据包括RNA序列,细胞流式分析,种丰富度,图像数据和单细胞测量等( RNA-Seq, flow-cytometry, taxa abundances, imaging data and single cell measurements)。作者贴心的说明学习本书不需要统计学背景(assume no prior training in statistics),但得了解一点R的知识,更重要的是愿意折腾——代码、公式、数学分析等。
第一章 广义模型和离散数据/Generative Models for Discrete Data
这一章先介绍简单模型的建立,但和刚才直接做个线性模型不同,本章从概率理论出发讲模型。概率和基因序列分析关系比较密切,结合免疫和DNA分析例子,介绍二项式/binomial, 多项式/multinomial 和泊松随机分布(变量)/ Poisson random variables。第二章 统计建模 Statistical Modeling
本章和上一章的关系在于,上一章看特定模型下数据长得什么样,这一章给定数据,看能应用什么模型,这才是建模的常规工作。第三章 R语言高质量作图 High Quality Graphics in R
讲了ggplot2,还有一些数据转化、作图语法方面的内容。第四章 混合模型 Mixture Models
数据肯定不会都顺顺当当的按模式分布,要刻画真实的数据,要把简单的模型进行组合,做成混合模型来反映实际。也包含一些数据转化的内容,这里转化要注意的是保证信息的稳定。(appropriate variance stabilizing transformations)第五章 聚类 Clustering
The large, matrix-like ( ) datasets in biology naturally lend themselves to clustering。数据只要能计算出Matrix-like的距离来,都可以应用聚类,这一章可能会大量介绍如何对不同类型的数据计算距离吧。第七章 多元统计 Multivariate Analysis
第九章 针对乱糟糟数据的多元方法 Multivariate methods for heterogeneous data
从逻辑上说,这两章虽然不挨着,但都是所谓EDA的范畴,也属于data discovery models或 unsupervised models。 第七章介绍了以主成分分析(PCA)为主线的多元统计方法,第九章深入应用这些方法,涉及非线性、分类变量、同一观测多重测定(multiple assays recorded on the same observational units)等第六章 检验 testing
检验可以针对假说、可以对模型,验证其是否合理,是否显著。介绍中说会实际检验一个 的数据集中,哪个基因或性状与某种疾病或其他表现是有联系的。但传统的显著性指标(也就是p值)不好使,所以要好好研究下多元检验(multiple testing)。第八章 高通量计数数据 High-Throughput Count Data
统计学一大利器是方差分解(variance decomposition),或曰方差分析(ANOVA)。本章以线性模型和广义线性模型的套路,分析RNA序列数据。分析也会涉及计数数据以及对稳健性(robustness)的讨论。第十章 网和树 Networks and Trees
据说生命科学中除了从进化的角度分析问题,其他啥都没多少意义。进化关系在系统发育树中体现,本章探索网和树的应用。第十一章 图像数据 Image data
在图像中提取特征数据,并进行分析,也是EDA的范畴。本章学习图像数据读写操作,分析方法等。第十二章 监督式学习 Supervised Leaning
属于统计学习的范畴(不知道算不算机器学习),如训练一个公式,基于某些多维特征向量分辨不同对象。从简单低维特征向量和线性方法开始,也探索高纬度分类设计。囧。。。说的啥啊这是。第十三章 高通量实验设计及分析 Design of High Throughput Experiments and their Analyses
介绍好的实验设计和相关数据分析,会对书中所学方法综合运用。
1.6 需要的计算机技能
通过对原书内容的概览,可以发现分析对象、角度、方法多种多样,要满足需求,要用 R 和 Bioconductor。
R是一种语言,一种为统计而生的语言;Bioconductor是基于R语言项目,专门提供搞生物数据的工具。要开始学习R语言有海量的资源,原书作者建议从 R for Data Science 开始学习,我也这么建议,因为Hadley确实是大神。 原书中用到的ggplot2, dplyr包,都是他写的。
另一个大神大家也可以关注,是中国人,现在美国,人大毕业,Rstudio主力程序员——谢益辉。作图和排版用的很多包,blogdown,bookdown,knitr等,都是他写的。包括本人现在写的这个记录,也是用bookdown写的,我的个人网站也是用的他的模板。
从这样的学科头部学者身上学习,会更有高度,更系统,当然也会更有难度,更需要学习能力。
Modern Statistics for Modern Biology的作者也是学科头部学者,而且这本书把数据代码都给出来,便于练习,是很好的学习材料。
References
Grolemund, Garrett, and Hadley Wickham. 2019. R for Data Science. https://r4ds.had.co.nz/.
Hastie, Trevor, Robert Tibshirani, and Jerome Friedman. 2016. The Elements of Statistical Learning: Data Mining, Inference, and Prediction, Second Edition. 2nd edition. New York, NY: Springer.
Karageorgiou, Elissaios. 2011. “The Logic of Exploratory and Confirmatory Data Analysis.” Cognitive Critique, January, 35–48.
Speed, Terry. 2011. “Large-Scale Inference: Empirical Bayes Methods for Estimation, Testing, and Prediction by Bradley Efron.” International Statistical Review 79 (1): 126–27. https://doi.org/10.1111/j.1751-5823.2011.00134_13.x.
Tukey, John W. 1977. Exploratory Data Analysis. Addison-Wesley Pub. Co.